Журнал «Здоровье ребенка» Том 17, №6, 2022
Вернуться к номеру
Механізми дії цитоплазматичних мікроРНК. Частина 5. МікроРНК-опосередкований сайленсинг, що викликається в періоді ініціації і постініціації трансляції
Авторы: Абатуров О.Є., Бабич В.Л.
Дніпровський державний медичний університет, м. Дніпро, Україна
Рубрики: Педиатрия/Неонатология
Разделы: Справочник специалиста
Версия для печати
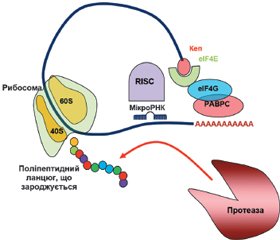
У науковому огляді подано механізми дії цитоплазматичних мікроРНК, а саме мікроРНК-опосередкований сайленсинг, що викликається в періоді ініціації і постініціації трансляції. Для написання статті здійснювався пошук інформації з використанням баз даних Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. Відомо, що мікроРНК-опосередкований сайленсинг, що викликається в періоді ініціації трансляції, відбувається за рахунок протеїнів Argonaute, які конкурують з кеп-зв’язуючими протеїнами й еукаріотичним фактором ініціації трансляції eIF4E під час взаємодії з 5’кеп-структурою мРНК. У кеп-залежній трансляції еукаріотичний фактор ініціації eIF4E розпізнає 5’кеп і сприяє рекрутингу інших факторів ініціації, зокрема eIF4G, для складання комплексу ініціації трансляції. Також фактор eIF4G взаємодіє з деякими протеїнами PABP, що призводить до утворення замкнутої петлі мРНК, обумовлюючи рекрутинг рибосоми. Наведено, що в періоді постініціації трансляції мікроРНК можуть: 1) викликати припинення трансляції, перешкоджаючи приєднанню або сприяючи дисоціації субодиниць рибосоми; 2) індукувати деградацію мРНК у періоді елонгації або 3) активувати деградацію і секвестрацію протеїнів. Автори показують, що мікроРНК можуть прямо або опосередковано пригнічувати функціонування рибосом, порушуючи формування компетентної 80S рибосоми, або перешкоджаючи приєднанню субодиниць рибосоми до мРНК або її просуванню по мРНК, або сприяючи дисоціації субодиниць рибосоми. Провідну роль у розвитку сайленсингу, обумовленого порушенням асоціації рибосомних субодиниць, відіграє протеїн AGO2. Автори показали, що комплекс «мікроРНК — мРНК-мішень» мігрує на більш легкі полісоми, ніж мРНК, яка не пов’язана з мікроРНК. Комплекс miRISC із мРНК і рибосомами може рекрутувати протеолітичні ферменти, які деградують виникаючий поліпептидний ланцюг. Отже, мікроРНК-опосередкований сайленсинг може викликатися в періодах ініціації і постініціації трансляції.
The scientific review considers the mechanisms of action of cytoplasmic microRNAs, namely miRNA-mediated silencing, which is caused during the initiation and post-initiation period of translation. To write the article, information was searched using Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka databases. It is known that miRNA-mediated silencing caused during translation initiation occurs due to Argonaute proteins, which compete with cap-binding proteins and the eukaryotic translation initiation factor eIF4E during interaction with the 5’cap structure of mRNA. In cap-dependent translation, the eukaryotic initiation factor eIF4E recognizes the 5’cap and promotes the recruitment of other initiation factors, in particular eIF4G, to assemble the translation initiation complex. Also, the eIF4G factor interacts with some PABP proteins, which leads to the formation of a closed loop of mRNA, determining the recruitment of the ribosome. It is stated that in the post-initiation period of translation, microRNAs can: 1) terminate translation, preventing the attachment or promoting the dissociation of ribosome subunits; 2) induce mRNA degradation during the elongation period or 3) activate protein degradation and sequestration. The authors state that microRNAs can directly or indirectly inhibit the functioning of ribosomes, disrupting the formation of a competent 80S ribosome, or preventing the attachment of ribosome subunits to mRNA, or its promotion along the mRNA, or promoting the dissociation of ribosome subunits. AGO2 protein plays a leading role in the development of silencing caused by disruption of the association of ribosomal subunits. The authors showed that the miRNA-mRNA-target complex migrates to lighter polysomes than mRNA that is not associated with miRNA. The miRISC complex with mRNA and ribosomes can recruit proteolytic enzymes that degrade the nascent polypeptide chain. Thus, miRNA-mediated silencing can be induced during the initiation and post-initiation periods of translation.
мікроРНК; мікроРНК-опосередкований сайленсинг; період ініціації трансляції; період постініціації трансляції; огляд
microRNA; miRNA; miR; miRNA-mediated silencing; initiation period of translation; post-initiation period of translation; review
Вступ
МікроРНК-опосередкований сайленсинг, що викликається в періоді ініціації трансляції
/53.jpg)
МікроРНК-опосередкований сайленсинг, що викликається в періоді постініціації трансляції
/55.jpg)
Висновки
- Aguilar-Valles A., Haji N., De Gregorio D. et al. Translational control of depression-like behavior via phosphorylation of eukaryotic translation initiation factor 4E. Nat. Commun. 2018. 9. 2459. https://doi.org/10.1038/s41467-018-04883-5.
- Allen G., Weiss B., Panasenk, O.O., Huch S., Villanyi Z., Albert B. et al. Not1 and Not4 inversely determine mRNA solubility that sets the dynamics of co-translational events. bioRxiv. 2022. https://doi.org/10.1101/2022.03.14.484207.
- Brina D., Grosso S., Miluzio A., Biffo S. Translational control by 80S formation and 60S availability: the central role of eIF6, a rate limiting factor in cell cycle progression and tumorigenesis. Cell. Cycle. 2011 Oct 15. 10(20). 3441-6. doi: 10.4161/cc.10.20.17796.
- Chendrimada T.P., Finn K.J., Ji X. et al. MicroRNA silencing through RISC recruitment of eIF6. Nature. 2007 Jun 14. 447(7146). 823-8. doi: 10.1038/nature05841.
- Christie M., Igreja C. eIF4E-homologous protein (4EHP): a multifarious cap-binding protein. The FEBS Journal. 2021. https://doi.org/10.1111/febs.16275.
- Da Sacco L., Masotti A. Recent insights and novel bioinformatics tools to understand the role of microRNAs binding to 5’ untranslated region. Int. J. Mol. Sci. 2012 Dec 27. 14(1). 480-95. doi: 10.3390/ijms14010480.
- Eulalio A., Huntzinger E., Izaurralde E. GW182 interaction with Argonaute is essential for miRNA-mediated translational repression and mRNA decay. Nat. Struct. Mol. Biol. 2008 Apr. 15(4). 346-53. doi: 10.1038/nsmb.1405.
- Fraser C.S. Quantitative studies of mRNA recruitment to the eukaryotic ribosome. Biochimie. 2015 Jul. 114. 58-71. doi: 10.1016/j.biochi.2015.02.017.
- Hendrickson D.G., Hogan D.J., McCullough H.L. et al. Concordant regulation of translation and mRNA abundance for hundreds of targets of a human microRNA. PLoS Biol. 2009 Nov. 7(11). e1000238. doi: 10.1371/journal.pbio.1000238.
- Hinnebusch A.G. Molecular mechanism of scanning and start codon selection in eukaryotes. Microbiol. Mol. Biol. Rev. 2011 Sep. 75(3). 434-67, first page of table of contents. doi: 10.1128/MMBR.00008-11.
- Hinnebusch A.G. The scanning mechanism of eukaryotic translation initiation. Annu Rev. Biochem. 2014. 83. 779-812. doi: 10.1146/annurev-biochem-060713-035802.
- Miluzio A., Beugnet A., Volta V., Biffo S. Eukaryotic initiation factor 6 mediates a continuum between 60S ribosome biogenesis and translation. EMBO Rep. 2009 May. 10(5). 459-65. doi: 10.1038/embor.2009.70.
- Mugridge J.S., Coller J., Gross J.D. Structural and molecular mechanisms for the control of eukaryotic 5’-3’ mRNA decay. Nat. Struct. Mol. Biol. 2018. 25. 1077-1085. https://doi.org/10.1038/s41594-018-0164-z.
- Petersen C.P., Bordeleau M.E., Pelletier J., Sharp P.A. Short RNAs repress translation after initiation in mammalian cells. Mol. Cell. 2006 Feb 17. 21(4). 533-42. doi: 10.1016/j.molcel.2006.01.031.
- Pillai R.S., Bhattacharyya S.N., Filipowicz W. Repression of protein synthesis by miRNAs: how many mechanisms? Trends Cell. Biol. 2007 Mar. 17(3). 118-26. doi: 10.1016/j.tcb.2006.12.007.
- Piserà A., Campo A., Campo S. Structure and functions of the translation initiation factor eIF4E and its role in cancer development and treatment. J. Genet. Genomics. 2018 Jan 20. 45(1). 13-24. doi: 10.1016/j.jgg.2018.01.003.
- Rhoads R.E. eIF4E: new family members, new binding partners, new roles. J. Biol. Chem. 2009 Jun 19. 284(25). 16711-5. doi: 10.1074/jbc.R900002200.
- Romagnoli A., D’Agostino M., Ardiccioni C. et al. Control of the eIF4E activity: structural insights and pharmacological implications. Cell. Mol. Life Sci. 2021. 78. 6869-6885. https://doi.org/10.1007/s00018-021-03938-z.
- Shirokikh N.E., Preiss T. Translation initiation by cap-dependent ribosome recruitment: Recent insights and open questions. Wiley Interdiscip. Rev. RNA. 2018 Jul. 9(4). e1473. doi: 10.1002/wrna.1473.
- Standart N., Jackson R.J. MicroRNAs repress translation of m7Gppp-capped target mRNAs in vitro by inhibiting initiation and promoting deadenylation. Genes. Dev. 2007 Aug 15. 21(16). 1975-82. Doi: 10.1101/gad.1591507.
- Takegaki J., Ogasawara R., Kouzaki K. et al. The distribution of eukaryotic initiation factor 4E after bouts of resistance exercise is altered by shortening of recovery periods. J. Physiol. Sci. 2020. 70. 54. https://doi.org/10.1186/s12576-020-00781-y.
- Tat T.T., Maroney P.A., Chamnongpol S. et al. Cotranslational microRNA mediated messenger RNA destabilization. Elife. 2016 Apr 8. 5. pii: e12880. doi: 10.7554/eLife.12880.
- Valinezhad Orang A., Safaralizadeh R., Kazemzadeh-Bavili M. Mechanisms of miRNA-Mediated Gene Regulation from Common Downregulation to mRNA-Specific Upregulation. Int. J. Genomics. 2014. 2014. 970607. doi: 10.1155/2014/970607.
- Virtue A., Wang H., Yang X.F. MicroRNAs and toll-like receptor/interleukin-1 receptor signaling. J. Hematol. Oncol. 2012 Oct 18. 5. 66. doi: 10.1186/1756-8722-5-66.
- Wang B., Yanez A., Novina C.D. MicroRNA-repressed mRNAs contain 40S but not 60S components. Proc. Natl Acad. Sci USA. 2008 Apr 8. 105(14). 5343-8. doi: 10.1073/pnas.0801102105.
- Wu C.Ch.-Ch., Zinshteyn B., Wehner K.A., Green R. High-Resolution Ribosome Profiling Defines Discrete Ribosome Elongation States and Translational Regulation during Cellular Stress. Molecular. Cell. 2019. 73(5). 959-970. e5. https://doi.org/10.1016/j.molcel.2018.12.009.

/54.jpg)