Международный эндокринологический журнал Том 19, №2, 2023
Вернуться к номеру
Оцінка впливу однонуклеотидних поліморфізмів генів рецептора вітаміну D (rs2228570), BDNF (rs6265) та NMDA (rs4880213) на експресію генів у різних тканинах
Авторы: I. Kamyshna (1), L. Pavlovych (2), I. Pankiv (2), V. Pankiv (3), A. Kamyshnyi (1)
(1) — I. Horbachevsky Ternopil National Medical University, Ternopil, Ukraine
(2) — Bukovinian State Medical University, Chernivtsi, Ukraine
(3) — Ukrainian Scientific and Practical Center for Endocrine Surgery, Transplantation of Endocrine Organs and Tissues of the Ministry of Health of Ukraine, Kyiv, Ukraine
Рубрики: Эндокринология
Разделы: Клинические исследования
Версия для печати
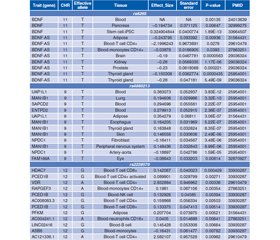
Актуальність. Питання щодо асоціації окремих та комбінованих варіацій і мутацій генів з хворобами щитоподібної залози та порушеннями нервової системи залишаються недостатньо дослідженими. Важливим аспектом є виявлення впливу окремих поліморфізмів генів на функціональну активність клітин, зокрема на експресію генів. На сьогодні генетика експресії генів значною мірою залежить від ідентифікації локусів кількісних ознак експресії (eQTL). Метою дослідження був пошук eQTL для однонуклеотидних поліморфізмів (SNP) генів BDNF (rs6265), VDR (rs2228570) та NMDA (rs4880213). Матеріали та методи. Використовували публічні бази даних (QTLbase: http://www.mulinlab.org/qtlbase/index.html, GTExPortal: https://gtexportal.org). Результати представлені у вигляді номінальних рівнів значущості для кожного SNP генів BDNF, VDR та NMDA. Результати. Використовуючи базу даних QTLbase, ми виявили статистично значущі (p ≤ 0,05) асоціації rs6265 з експресію 17 генів (BDNF-AS, BDNF, LDHC, AC104563.1, BBOX1, SPTY2D1OS, YWHABP2, LINC00678, LIN7C, GTF2H1, METTL15, IMMP1L, KIF18A, HPS5, NAV2, LGR4, CCDC34) у різних тканинах. Для rs4880213 ми знайшли асоціації з рівнями експресії 49 генів (ARRDC1-AS1, TPRN, SSNA1, SAPCD2, UAP1L1, NPDC1, MAN1B1, PTGDS, SNHG7, NDOR1, TRAF2, PHPT1, EGFL7, EHMT1, RNF208, PNPLA7, LCNL1, DPP7, LCN12, STPG3, CCDC183-AS1, ABCA2, RNF224, ENTPD2, PAXX, CLIC3, C9orf163, LCN15, MAN1B1-DT, FAM166A, FAM166A, LRRC26, STPG3-AS1, AGPAT2, ANAPC2, DPH7, ZMYND19, NSMF, MRPL341, EXD, TUBB4B, NELFB, ARRDC1, EDF1, FBXW5, DIPK1B, MAMDC4, RABL6, TMEM141, TMEM203) у 16 різних тканинах. Крім того, виявили статистично значущі (p ≤ 0,05) зв’язки між rs2228570 та експресією 29 генів (ASB8, TMEM106C, KANSL2, DDX23, CCNT1, HDAC7, RPAP3, PFKM, SENP1, RND1, PCED1B, AC004466.1, AMIGO2, ZNF641, ENDOU, RAPGEF3, VDR, AC004241.1, AC004801.2, AC121338.1, LINC02354, SNORA2A, LINC02416, AC074029.3, AC004241.5, AC008083.3, COL2A1, CCDC184, SLC48A1) у 17 різних тканинах. Висновки. Однонуклеотидні поліморфізми генів BDNF (rs6265), VDR (rs2228570) і NMDA (rs4880213) впливають на експресію генів у різних клітинах і тканинах. Застосування каталога eQTL надає важливий ресурс для розуміння молекулярної основи поширених генетичних захворювань.
Background. Questions regarding the association of individual and combined gene variations and mutations with thyroid disease and nervous system disorders remain insufficiently researched and require further study to facilitate early diagnosis of nervous system damage on the background of thyroid pathology, disease prognosis, and timely treatment and prevention. An important issue is the identification of the influence of individual polymorphisms in these genes on the functional activity of cells, including gene expression. Currently, gene expression genetics largely depends on the identification of expression quantitative trait loci (eQTL), which are the links between gene expression and genotype at a locus. The purpose of the study was to search for eQTL in single nucleotide polymorphisms (SNPs) of the BDNF gene (rs6265), VDR gene (rs2228570), and NMDA gene (rs4880213). The results were presented as nominal p-values for each SNP of the BDNF, VDR, and NMDA genes. Materials and methods. We use publicly available databases (QTLbase: http://www.mulinlab.org/qtlbase/index.html, GTExPortal: https://gtexportal.org). Results. Using the QTLbase, we identified statistically significant (p ≤ 0.05) associations of rs6265 with the expression of 17 genes (BDNF-AS, BDNF, LDHC, AC104563.1, BBOX1, SPTY2D1OS, YWHABP2, LINC00678, LIN7C, GTF2H1, METTL15, IMMP1L, KIF18A, HPS5, NAV2, LGR4, CCDC34) in various tissues. For rs4880213, we found a significant association with the expression levels of 49 genes (ARRDC1-AS1, TPRN, SSNA1, SAPCD2, UAP1L1, NPDC1, MAN1B1, PTGDS, SNHG7, NDOR1, TRAF2, PHPT1, EGFL7, EHMT1, RNF208, PNPLA7, LCNL1, DPP7, LCN12, STPG3, CCDC183-AS1, ABCA2, RNF224, ENTPD2, PAXX, CLIC3, C9orf163, LCN15, MAN1B1-DT, FAM166A, FAM166A, LRRC26, STPG3-AS1, AGPAT2, ANAPC2, DPH7, ZMYND19, NSMF, MRPL41, EXD3, TUBB4B, NELFB, ARRDC1, EDF1, FBXW5, DIPK1B, MAMDC4, RABL6, TMEM141, TMEM203) in 16 different tissues. Additionally, we identified statistically significant (p ≤ 0.05) associations of rs2228570 with the expression of 29 genes (ASB8, TMEM106C, KANSL2, DDX23, CCNT1, HDAC7, RPAP3, PFKM, SENP1, RND1, PCED1B, AC004466.1, AMIGO2, ZNF641, ENDOU, RAPGEF3, VDR, AC004241.1, AC004801.2, AC121338.1, LINC02354, SNORA2A, LINC02416, AC074029.3, AC004241.5, AC008083.3, COL2A1, CCDC184, SLC48A1) in 17 different tissues. Conclusions. Single nucleotide polymorphisms of the BDNF (rs6265), VDR (rs2228570), and NMDA genes (rs4880213) affect gene expression in various cells and tissues. The use of this extensive eQTL catalog provides an important resource for understanding the molecular basis of common genetic diseases.
генотип; експресія; поліморфізм нуклеотидів; ген BDNF (rs6265); ген VDR (rs2228570); ген NMDA (rs4880213)
genotype; expression; nucleotide polymorphisms; BDNF gene (rs6265); VDR gene (rs2228570); NMDA gene (rs4880213)
Для ознакомления с полным содержанием статьи необходимо оформить подписку на журнал.
- Cockrum C., Kaneshiro K.R., Rechtsteiner A., Tabuchi T.M., Strome S. A primer for generating and using transcriptome data and gene sets. Development. 2020 Dec 23. 147(24). dev193854. doi: 10.1242/dev.193854.
- Liu L., Tamura K., Sanderford M., Gray V.E., Kumar S. A Molecular Evolutionary Reference for the Human Variome. Mol. Biol. Evol. 2016 Jan. 33(1). 245-54. doi: 10.1093/molbev/msv198.
- Vihinen M., Hancock J.M., Maglott D.R., Landrum M.J., Schaafsma G.C., Taschner P. Human Variome Project Quality Assessment Criteria for Variation Databases. Hum. Mutat. 2016 Jun. 37(6). 549-58. doi: 10.1002/humu.22976.
- Thul P.J., Åkesson L., Wiking M., Mahdessian D., Geladaki A., Ait Blal H., Alm T. et al. A subcellular map of the human proteome. Science. 2017 May 26. 356(6340). eaal3321. doi: 10.1126/science.aal3321.
- Dzhuryak V., Sydorchuk L., Sydorchuk A., Kamyshnyi O., Kshanovska A., Levytska S. et al. The cytochrome 11B2 aldosterone synthase gene CYP11B2 (RS1799998) polymorphism associates with chronic kidney disease in hypertensive patients. Biointerface Res. Appl. Chem. 2020. 10(3). 5406-11. doi: 10.33263/BRIAC103.406411.
- Kamyshna I., Pavlovych L., Pankiv I., Pankiv V., Maslyanko V., Bytsko N., Kamyshnyi A. The complex influence of the combination of the BDNF (rs6265), VDR (rs2228570), and NMDA (rs4880213) genotypes on the development of cognitive disorders in patients with autoimmune thyroiditis and hypothyroidism. International Journal of Endocrinology (Ukraine). 2023. 19(1). 9-15. doi: 10.22141/2224-0721.19.1.2023.1235.
- Bankier S., Michoel T. eQTLs as causal instruments for the reconstruction of hormone linked gene networks. Front. Endocrinol. (Lausanne). 2022 Aug 17. 13. 949061. doi: 10.3389/fendo.2022.949061.
- Wen J., Nodzak C., Shi X. QTL Analysis Beyond eQTLs. Methods Mol. Biol. 2020. 2082. 201-210. doi: 10.1007/978-1-0716-0026-9_14.
- Yang S., Liu Y., Jiang N., Chen J., Leach L., Luo Z., Wang M. Genome-wide eQTLs and heritability for gene expression traits in unrelated individuals. BMC Genomics. 2014 Jan 9. 15(1). 13. doi: 10.1186/1471-2164-15-13.
- Kamyshna I.I., Pavlovych L.B., Malyk I.V., Kamyshnyi A.M. 25-OH Vitamin D blood serum linkage with VDR gene polymorphism (rs2228570) in thyroid pathology patients in the West-Ukrainian po–pulation. J. Med. Life. 2021 Jul-Aug. 14(4). 549-556. doi: 10.25122/jml-2021-0101.
- Nica A.C., Dermitzakis E.T. Expression quantitative trait loci: present and future. Philos. Trans. R. Soc. Lond. B Biol. Sci. 2013 May 6. 368(1620). 20120362. doi: 10.1098/rstb.2012.0362.
- Lee C. Genome-Wide Expression Quantitative Trait Loci Analysis Using Mixed Models. Front. Genet. 2018 Aug 21. 9. 341. doi: 10.3389/fgene.2018.00341.
- Quiver M.H., Lachance J. Adaptive eQTLs reveal the evolutionary impacts of pleiotropy and tissue-specificity while contributing to health and disease. HGG Adv. 2021 Dec 24. 3(1). 100083. doi: 10.1016/j.xhgg.2021.100083.
- Shen F., Gan X., Zhong R., Feng J., Chen Z., Guo M., Li Y. et al. Identifying Thyroid Carcinoma-Related Genes by Integrating GWAS and eQTL Data. Front. Cell. Dev. Biol. 2021 Feb 4. 9. 645275. doi: 10.3389/fcell.2021.645275.
- Yang H., Liu D., Zhao C., Feng B., Lu W., Yang X., Xu M., Zhou W., Jing H., Yang J. Mendelian randomization integrating GWAS and eQTL data revealed genes pleiotropically associated with major depressive disorder. Transl. Psychiatry. 2021 Apr 17. 11(1). 225. doi: 10.1038/s41398-021-01348-0.
- Aman A., Slob E.A.W., Ward J., Cullen B., Graham N., Lyall D.M., Sattar N., Strawbridge R.J. Investigating the potential impact of PCSK9-inhibitors on mood disorders using eQTL-based Mendelian randomization. PLoS One. 2022 Dec 29. 17(12). e0279381. doi: 10.1371/journal.pone.0279381.